En la procezo de farado de genetikaj studoj, ni ofte renkontas nesufiĉajn RNA-provaĵojn, ekzemple, por studi etajn anatomiajn buŝajn tumorojn, eĉ unuĉelaj specimenoj, kaj provaĵoj de specifaj genmutacioj kiuj estas transskribitaj ĉe tre malaltaj niveloj en homaj ĉeloj.Kompreneble, por la testo COVID-19, se la lamboj ne estas en la ĝusta loko aŭ ne sufiĉe da tempoj dum specimenigo, la specimena grandeco estos tre malalta, tial la Komisiono pri Sano kaj Familia Planado aperis antaŭ du tagoj kaj pasigis la teston, kaj se la nukleacida samplilo ne prenis ses specimenojn, vi povas raporti ĝin.
La sentemo de la reakciilo estas grava ĉar ni havas ĉi tiun aŭ tiun problemon, do kion ni povas fari por plibonigi la sentemon de RT-PCR?
Antaŭ ol ni diskutas eblajn solvojn, ni menciu du grandajn komplikaĵojn kun la situacio, kiun ni ĵus menciis.
Antaŭ ĉio, ni zorgas pri RNA-perdo kiam ni havas nur kelkajn ĉelpopulaciojn en nia specimeno.Se oni uzas tradiciajn disigajn kaj purigajn metodojn, kiel kolumnan metodon aŭ nukleacidan precipitan metodon, estas alta ebleco, ke la malmultaj specimenoj perdiĝos.Unu solvo estas aldoni portantan molekulon, kiel tRNA, sed eĉ tiam ne estas garantio, ke nia reakira eksperimento estas bona.
Do kio estas pli bona maniero?Bona elekto por kulturitaj ĉeloj aŭ mikroanatomiaj specimenoj estas uzi rektan lizon.
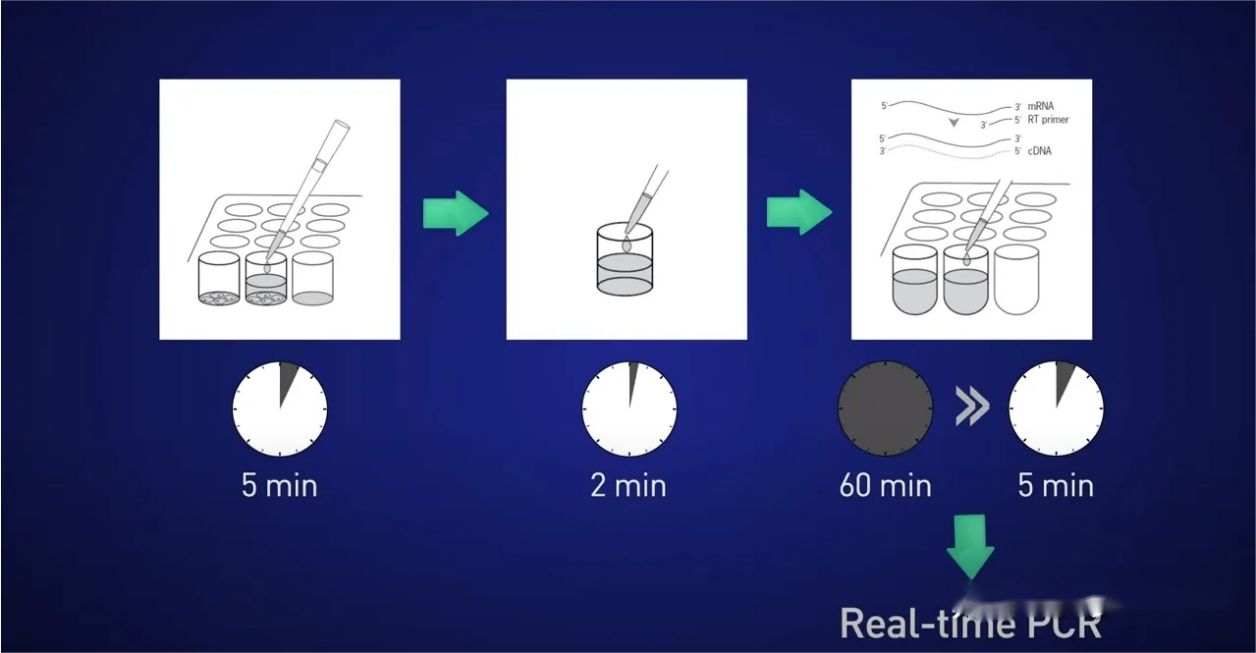
La ideo estas dividi la ĉelojn dum 5 minutoj, liberigi la RNA en la solvaĵon, poste ĉesigi la reagon dum 2 minutoj, poste aldoni la lisaton rekte al la inversa transskriba reago por ke neniu RNA perdiĝu, kaj fine rekte meti la rezultan cDNA. en la realtempan reagon.
Sed kio se, pro limigita deirpunkto aŭ malgranda kvanto de cela genesprimo, ni povas recikli la tutan RNA kaj ankoraŭ ne provizi sufiĉajn ŝablonojn por ricevi bonan realtempan signalon?
En ĉi tiu kazo, la antaŭ-amplifiga paŝo povas esti tre utila.
La sekvanta estas skemo por pliigi sentemon post inversa transskribo.Antaŭ ol komenci, ni devas demandi laŭflue, pri kiuj celoj ni interesiĝas, por desegni specifajn enkondukojn por ĉi tiuj celoj por antaŭ-plifortigo.
Ĉi tio povas esti atingita kreante miksitan enkondukon kun ĝis 100 paroj da enkondukoj kaj reagciklo de 10 ĝis 14 fojojn.Tial, Majstra Miksaĵo specife desegnita por ĉi tiu postulo estas necesa por antaŭ-amplifi la cDNA akiritan.
La kialo por fiksi la nombron da cikloj inter 10 kaj 14 estas, ke ĉi tiu limigita nombro da cikloj certigas hazardon inter la diversaj celoj, kio estas decida por esploristoj, kiuj bezonas kvantajn molekulajn informojn.
Post antaŭ-amplificado, ni povas akiri grandan kvanton da cDNA, tiel ke la detekta sentemo ĉe la malantaŭa fino estas multe plibonigita, kaj ni eĉ povas dilui la specimenon kaj plenumi plurajn realtempajn PCR-reagojn por forigi eblajn hazardajn erarojn.
Afiŝtempo: Apr-11-2023